继续更新一下基因组注释,在基因组注释的第5篇博客中,我们已经拿到了Braker3预测的功能基因,并且删除了其中可能被TE插入而失去功能的基因。仅仅拿到这些基因CDS序列和蛋白序列肯定是不够的,我们还需要知道这些蛋白具体行使什么生物学功能。
1. 功能注释常用数据库
基因功能注释的原理都是相似的,我们拿到了蛋白序列之后,与现有的蛋白数据库进行比对,统计比对结果。稍微介绍几个常用的比对数据库,其他没介绍到的可以参考本站网址导航—生信网站快速导航,里面有本人年初总结的一些常用生信数据库和工具(有好的工具也可以联系我补充)。
1.1 NR数据库
NR数据库全称Non-Redundant Protein Sequence Database(非冗余蛋白数据库),由NCBI建立和维护,可以在NCBI主页的Data&Software入口找到FTP:BLAST Databases。用过本地blast的人都知道,blast比对NR/NT库的结果中包含了物种注释信息和相应的氨基酸序列,可以用于物种分类、数据污染评估等等(没用过的话可以参考我的这篇博客——数据污染评估)。
一般的注释方法和数据污染评估一样,使用blastp的比对方法,设置-evalue 1e-5, -max_target_seqs 1,只保留匹配得分最高的蛋白序列。
1.2 KEGG数据库
KEGG数据库全称Kyoto Encyclopedia of Genes and Genomes,是日本京都大学生物信息学中心1995年建立的数据库,该数据库描述生物体中复杂的生物学通路,其丰富的通路信息帮助我们系统了解蛋白的生物学功能,如代谢通路、遗传信息传递以及细胞过程等一些复杂的生物功能。
一般的注释方法同上,比对的数据库为KEGG数据库,并统计蛋白序列注释到的KEGG通路信息。
1.3 KOG数据库
KOG数据库全称Eukaryotic Orthologous Groups of protein(真核同源群数据库),和这个数据库齐名的还有COG(Clusters of Orthologous Groups of proteins),这两个数据库都是NCBI中基于直系同源关系的数据库,COG针对原核生物,KOG针对真核生物。其比对原理是将蛋白序列比对并注释到某个同源蛋白簇中,这个同源蛋白簇是NCBI通过生物的完整基因组的编码蛋白系统进化关系分类构建而成的,比对到的同源蛋白簇揭示该蛋白的功能。
一般的注释方法同上,比对的是KOG数据库,并统计蛋白序列注释的KOG信息。
1.4 GO数据库
GO数据库全称Gene Ontology,数据库由基因本体联合会建立,是将所有与基因有关的研究结果进行分类汇总的综合数据库。可能听起来比较抽象,基因本体论的本质是用特定的一套词汇描述生物学功能,对不同物种的基因功能进行注释的统一化(去掉物种特异性),我们可以将基因按照其参与的生物过程(BiologicalProcess, BP) 、 细胞组分(Cellular Component, CC) , 分子功能(Molecular Function, MF) 三个方面进行分类注释。很多蛋白数据库都带有GO号注释信息。
GO注释目前有两种主要的注释方法:
- 序列相似性比对,也就是上面blast方法,比对的数据库可以是Swissport,提取GO列的注释信息。
- 结构域相似性比对,常用的软件有
InterproScan,比对的数据库可以是Pfam数据库(一个蛋白质域家族的数据库),也是提取GO注释信息。
1.5 Swissprot数据库
Swissprot(不要打成Swissport,瑞士机场)隶属于Uniprot数据库, 包含经过注释和验证的严格非冗余的蛋白序列数据库, 提供了蛋白序列详尽的注释信息。也是一个非常常用的蛋白注释数据库,现在UniprotKB(Universal Protein Knowledge Base)主要由两个子库构成,一个是TrEMB(该部分是计算机进行分析注释的,未人工校验的蛋白),另一个就是Swissprot。
一般的注释方法同上,比对Swissprot数据库,统计基因组蛋白序列注释到的Swissprot数据库蛋白信息。
上面介绍的是一般情况下我们做基因功能注释要用的数据库,全都是可以用blast比对相应的数据库,最后做个汇总比如韦恩图查看各个数据库注释的情况。
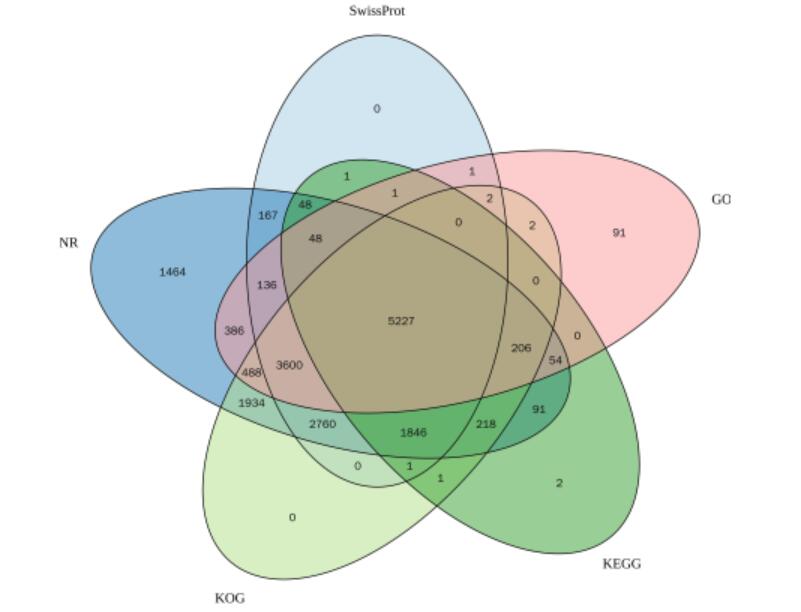
之所以说一般情况,是因为随着算法和技术的发展,不断有新的比对工具(比如DIAMOND,速度是blast的100到10000倍;还有基于隐马尔科夫模型的HMMER 3等)和注释流程的出现,这些经典的方法固然可用,缺点也很明显:比对速度慢,需要下载大量的比对数据库,占用资源等等。
2. eggNOG
eggNOG(evolutionary gene genealogy Non-supervised Orthologous Groups)数据库是从NCBI的COG/KOG延伸出来的,前面介绍过这两个数据库是直系同源数据库,在2008年提出的eggNOG的时候只有这两个直系同源数据库,这两个数据库在当时一方面是数据不多(当时只有 312 个细菌、26 个古细菌和 35 个真核生物基因组),另一方面是更新缓慢,主要对当时比较基因组学研究影响较大。
看到Non-supervised无监督这几个字,相信有的小伙伴就明白了,他们用了Smith–Waterman算法(与blast齐名的寻找序列最优相似比较的算法,比blast更精准)构建了直系同源群(Orthologous Group),并且通过基因的描述文件、注释的功能类别和预测的蛋白质结构域等,自动注释这些直系同源群。这种非监督的方法,不受物种限制,不限于已有基因的功能和关系,这种灵活性、可扩展性和跨物种比较的优势,可以帮助人们发现新的功能和关系,并提供基于进化关系的功能注释。
感兴趣的可以看看下面这篇2008年的原文:
eggNOG: automated construction and annotation of orthologous groups of genes - PMC (nih.gov)
经过十几年的迭代,eggNOG现在已经发布了6.0版本(2023年1月6日),总的来说,eggNOG 6.0提供了超过1700万个直系同源群 (OG) ,涵盖10756个细菌、457个古细菌和1322个真核生物,这些直系同源群可注释的信息包括 KEGG、GO、UniProtKB、BiGG、CAZy、CARD、PFAM 和 SMART。此外,eggNOG 6.0网站还推出了挖掘直系同源基因和基因功能数据分析的新功能,包括为跨物种的多个直系同源群生成系统发育图谱等。
3. eggNOG-mapper
eggNOG-mapper是eggNOG推出的一款进行批量基因功能注释的工具,在2021年的时候已经更新到了v2版本,主要更新了从raw contigs开始的从头基因预测、内置成对的同源预测(built-in pairwise orthology prediction)、快速检测蛋白结构域和自动生成gff文件四项功能。
3.1 eggNOG-mapper原理
整个流程和原理可以用作者原文的一张图概括:
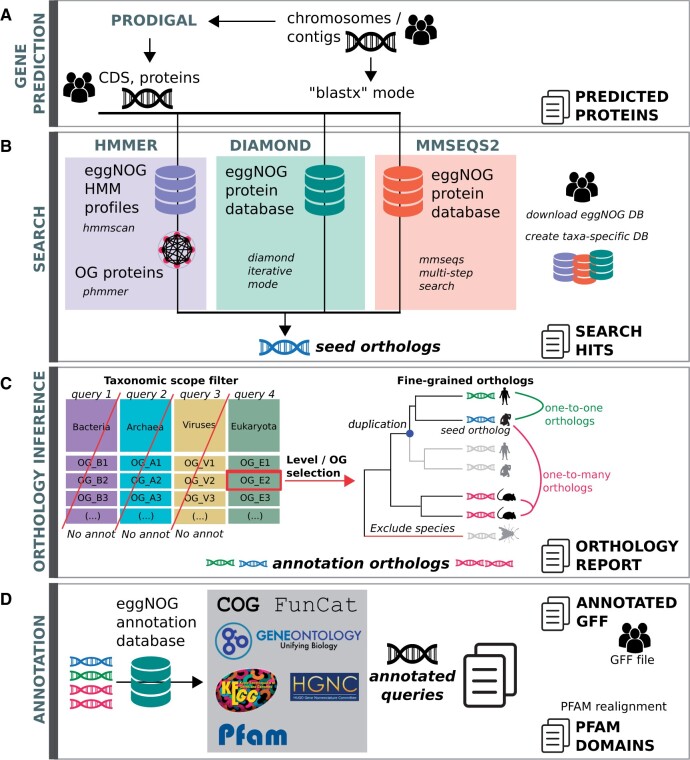
A: 编码基因预测阶段,用Prodigal从组装的contigs中进行蛋白预测(或者blastx模式预测)。
B: 搜索阶段,选择HMMER或者DIAMOND或者MMESQS2将输入的蛋白序列和EggNOG数据库进行比对,生成seed orthologs。
C: 直系同源推断阶段,根据所需要的分类范围,生成直系同源报告。
D: 注释阶段,根据蛋白注释信息和蛋白结构域注释信息,生成表格和gff格式的报告。
3.2 运行网页版eggNOG-mapper
eggNOG-mapper这个工具可以下载到本地运行(主要是方便宏基因组这种大批量的注释),也可以在线运行,官方提供了在线服务地址http://eggnog-mapper.embl.de
因为我自己不是做宏基因组的,所以也没必要下载工具和数据库(需要的时候再更),直接使用在线服务就可以。
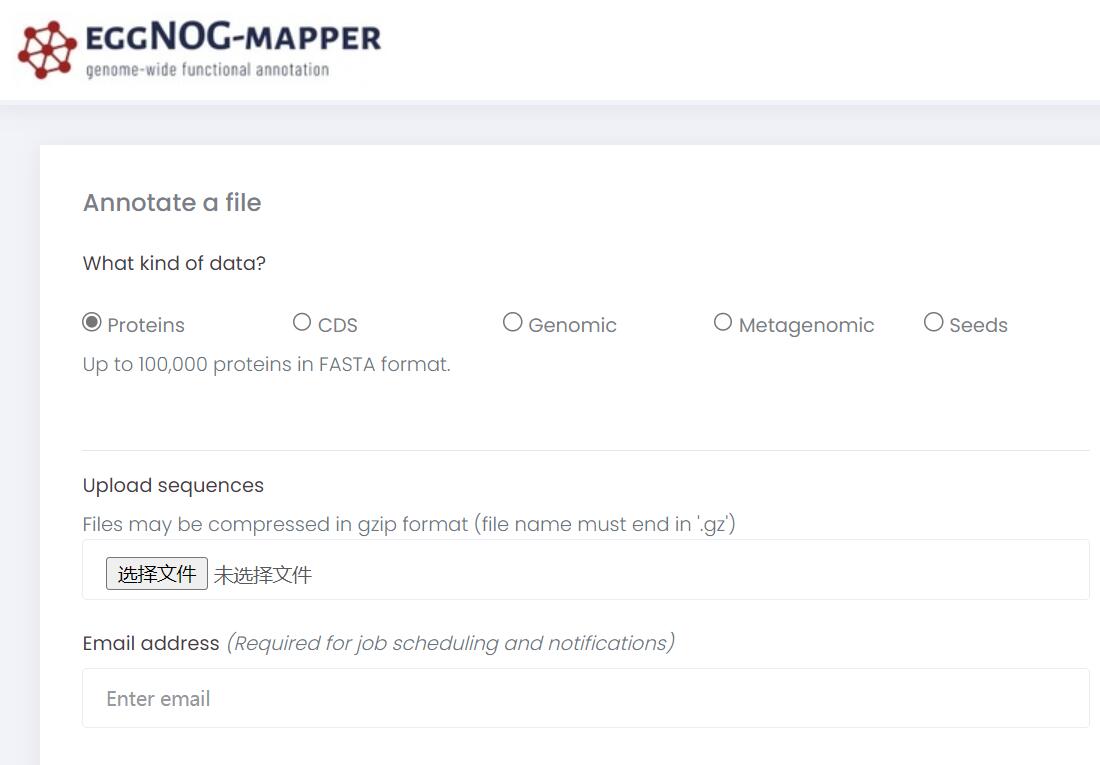
可以看到eggNOG-mapper在线版支持5种类型数据输入:
Proteins:蛋白序列,上限是10万条序列
CDS:CDS序列,上限也是10万条,会在搜索前倍翻译成蛋白序列
Genomic:基因组序列,支持最多1000条DNA序列,1000万个核苷酸。直接上传基因组序列会多一步编码蛋白预测,可以选择使用
Prodigal或者Blastx-like这两者之一。Metagenomic:Contig级别的基因组序列,本质上和上面是一样的,所以限制条件也相同。同样会进行编码蛋白预测。
Seeds:eggNOG-mapper跑的seed orthologs,支持最多上传10万条,这个主要用于重新注释。
我们前面拿到了蛋白序列,所以直接上传蛋白序列即可,注意一下上传的要求是.gz的压缩文件。所以稍微处理下:
1 | 保留原文件,以gz格式压缩为另一个文件 |
上传gz文件,留下邮箱,底下还可以设置比对的参数和注释的参数,都以默认参数运行即可。稍微说下注释的一些参数:
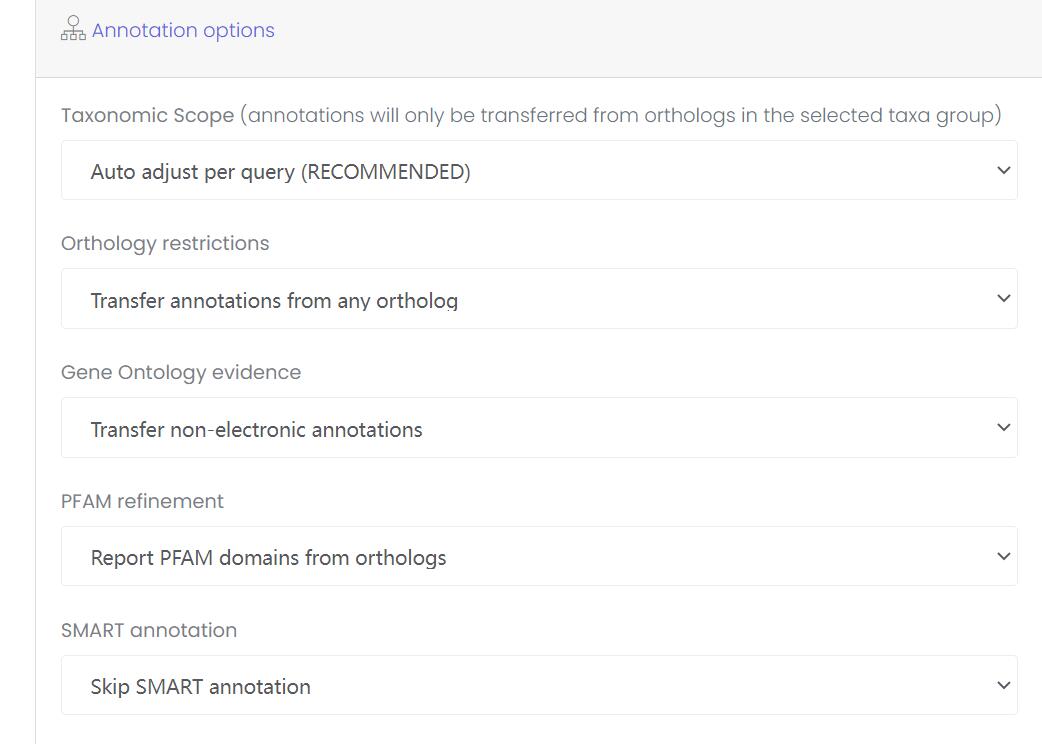
Taxonomic Scope:可以手动选择的分类范围,官方建议是默认的方式,但也可以手动选择,比如我注释植物,就可以选择Viridiplantae绿色植物。这里的参数会影响流程里的直系同源推断阶段的范围,默认情况下每个序列都会自动调整。
Orthology restrictions:直系同源限制,可以选择from any ortholog或者from one to one ortholog,也是影响直系同源推断阶段的结果,后者只会在直系同源中匹配。
Gene Ontology evidence:基因本体论证据,这里是两种:1. experimental,也就是有实验支撑的(比如做过亚细胞定位啊,免疫荧光啊这些)。2. non-electronic,通俗说就是“非电子”证据,电子证据是与实验证据相距最远的,这里的指的是除实验和电子以外的证据。
PFAM refinement:这里的选项是对蛋白结构域注释的调整,可以直接出报告,也可以再将序列进行realign等。
SMART annotation:顾名思义,就是这步可以用SMART再进行蛋白质结构域预测,默认是跳过的。
关于基因本体论的证据,GO官网有解释,我这里解释可能不是很清楚,可以查看官网对基因本体论的证据说明以及分类:
Guide to GO evidence codes (geneontology.org)
所有参数确定之后,点击submit,网页会提醒你查看邮件。进入邮箱,点击第一个第一个选项Click to manage your job,再点击Start job即可开始运行。

3.3 eggNOG-mapper结果文件解读
在线运行还是非常快的~我这20000个左右的蛋白序列,吃个饭的功夫回来就跑完了,不用下数据库不用配置环境就可以做分析,一个字,香!
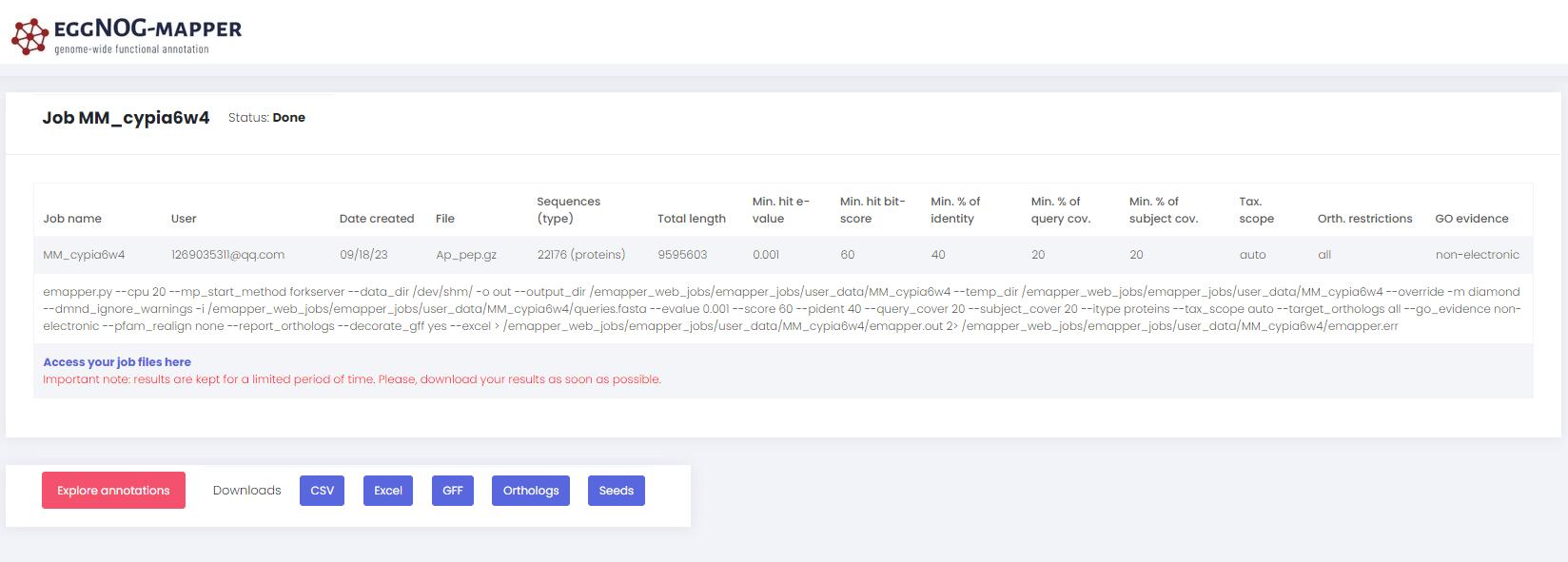
当我们看到右上角状态为Done后,就证明已经跑完了。我们返回前一个页面,点击Access your job files here,这个链接就是我们结果文件的存储位置,注意存储是暂时的,需要我们尽快下载到本地保存。
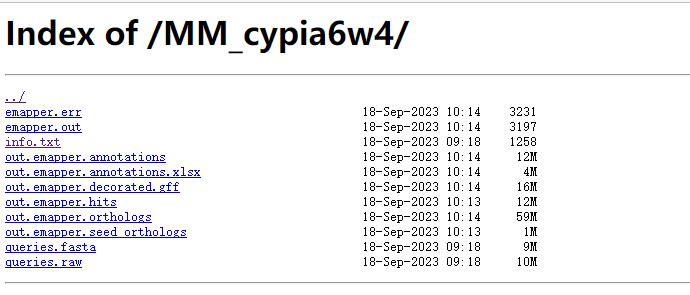
检查日志无报错即可。我们主要用的是其中的两个annotations文件,一个是方便你自己提取内容做富集分析(内容制表符分割,怎么提取和做GO、KEGG富集分析下次再说吧,写个脚本的事),第二个带.xlsx后缀方便你用excel编辑和打开。
这里主要讲一下结果文件的各项参数代表什么意思:

- query:蛋白序列名称
- seed_ortholog:搜索阶段比对上的seed ortholog编号
- evalue:evalue值,越小结果越可靠
- score:比对的得分,越大越可靠
- eggNOG_OGs:为序列确定的以逗号分隔、按照进化分支深度排序的直系同源组(orthologs groups,OGs)列表。每个直系同源组以OG@tax_id|tax_name方式展现。
- max_annot_lvl:用于检索注释的最宽的直系同源组,以tax_id|tax_name方式展现。
- COG_category:从最佳OG中推测的COG功能分类(一个字母),具体有哪些可以看这里NCBI的介绍COG - NCBI (nih.gov)。
- Description:注释的基因功能描述(eggNOG-mapper注释的描述真的很简短……)
- Preferred_name:普遍使用的基因名缩写
- GOs:注释的GO编号,一个基因可能对映非常多的GO号
- EC:KEGG的EC通路编号,代表相关的酶
- KEGG_ko:KEGG的KO编号,表示直系同源基因,代表一个具体的功能
- KEGG_Pathway:通常有ko编号和map编号,map编号代表reference pathway,是一种代谢通路类型,比较具体有一般参考意义
- KEGG_Module:KEGG Module数据库编号,以M开头,实际上就是多个KO划分在一个共同发挥功能的单元里
- KEGG_Reaction:KEGG Reaction数据库编号,以R开头,包含代谢通路上酶促反应相关信息
- KEGG_rclass:KEGG RCLASS数据库编号,以RC编号开头,手动整理的反应数据集合
- BRITE:KEGG的Brite数据库编号,用的不是很多,是一个储存分类信息的数据库
- CAZy:碳水化合物酶相关的专业数据库,可以看官网CAZy - Home
- BiGG_Reaction:BiGG是一个整合基因组尺度代谢网络模型的数据库,官网BiGG Models (ucsd.edu)
- PFAMs:PFAM数据库,根据多序列比对结果和隐马尔可夫模型,将蛋白分为不同家族的一个数据库,官网InterPro (ebi.ac.uk)。没错官网是InterPro,pfam数据库已经被InterPro合并,并且2023年1月之后原网页就失效了。
这么多数据咋一看很头疼,可以整理一下写个脚本,做GO和KEGG富集分析,这个下次再说。






